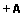Resumo: A estreita base genética da soja dificulta a caracterização das cultivares com base em marcadores morfológicos, principalmente para registro e proteção no Serviço Nacional de Proteção de Cultivares. Os marcadores moleculares têm se tornado uma ferramenta importante nos casos de indistinguibilidade de cultivares, pois não são influenciados pelo ambiente e podem ser utilizados em qualquer estádio de desenvolvimento da planta. Dentre os marcadores moleculares, os microssatélites estão distribuídos ao longo de todo genoma, são específicos, multialélicos e codominantes. O presente trabalho teve como objetivo caracterizar um conjunto de 48 cultivares de soja, utilizando marcadores microssatélites detectados em sequenciador automático; estimar as frequências alélicas de um conjunto de 24 marcadores microssatélites e trabalhar com um número mínimo de marcadores para a caracterização inequívoca. As análises dos fragmentos foram geradas em sequenciador automático ABI PRISM Genetic Analyser® 3130xl e o software Genotyper® foi aplicado para a visualização exata dos alelos e para emissão de dados automaticamente. Os eletroferogramas obtidos permitiram a identificação dos marcadores mais informativos e de seus respectivos alelos. No total foram observados 173 alelos, com uma média de 7,17 alelos por marcadores. Os marcadores mais polimórficos foram Sat_105 e GMABAB. Os marcadores Satt216, Satt586, Satt233, Satt181 e Satt070 foram capazes de identificar e diferenciar todas as 48 cultivares. Com base nesses marcadores foi possível a elaboração das etiquetas genéticas e a criação de um banco de dados precioso. Tal sistema poderá ser utilizado para a caracterização de novas cultivares, auxiliando na proteção.
Abstract: The narrow genetic base of soybean has made cultivars characterization based on morphological markers very difficult, mainly for register and protection at the National Service for Plant Variety Protection. Molecular markers have become an important tool as complementary information in cases of indistinguishable cultivars, once they are not environmental sensitive and can be applied at any stage of plant development. Among molecular markers, microsatellites are distributed through all plant genome, specific, multiallelic and co-dominant. This study had as objectives to characterize a group of 48 soybean cultivars using microsatellites molecular markers detected in automated sequencer; to estimate the allelic frequencies of 24 SSR and to estimate the minimum number of markers for unequivocal characterization. The analyses of amplified fragments were carried out in the automatic sequencer ABI PRISM Genetic Analyser® 3130xl and the GeneMapper® software was applied to precisely identify the alleles in an automatic fashion.The electropherograms allowed the identification of informative loci and its alleles. A total of 173 alleles were observed, with an average of 7.17 alleles per loci. The most informative markers were Sat_105 and GMABAB. The SSR markers Satt216, Satt586, Satt233, Satt181 and Satt070 were able to identify all 48 cultivars. Based on these loci it was possible to elaborate genetic tags and also to create a valuable microsatellites database. This system can be used in the characterization of new cultivars, assisting the protection procedure, as well as in cases of seed genetic purity when visual evaluations are doubtful. |